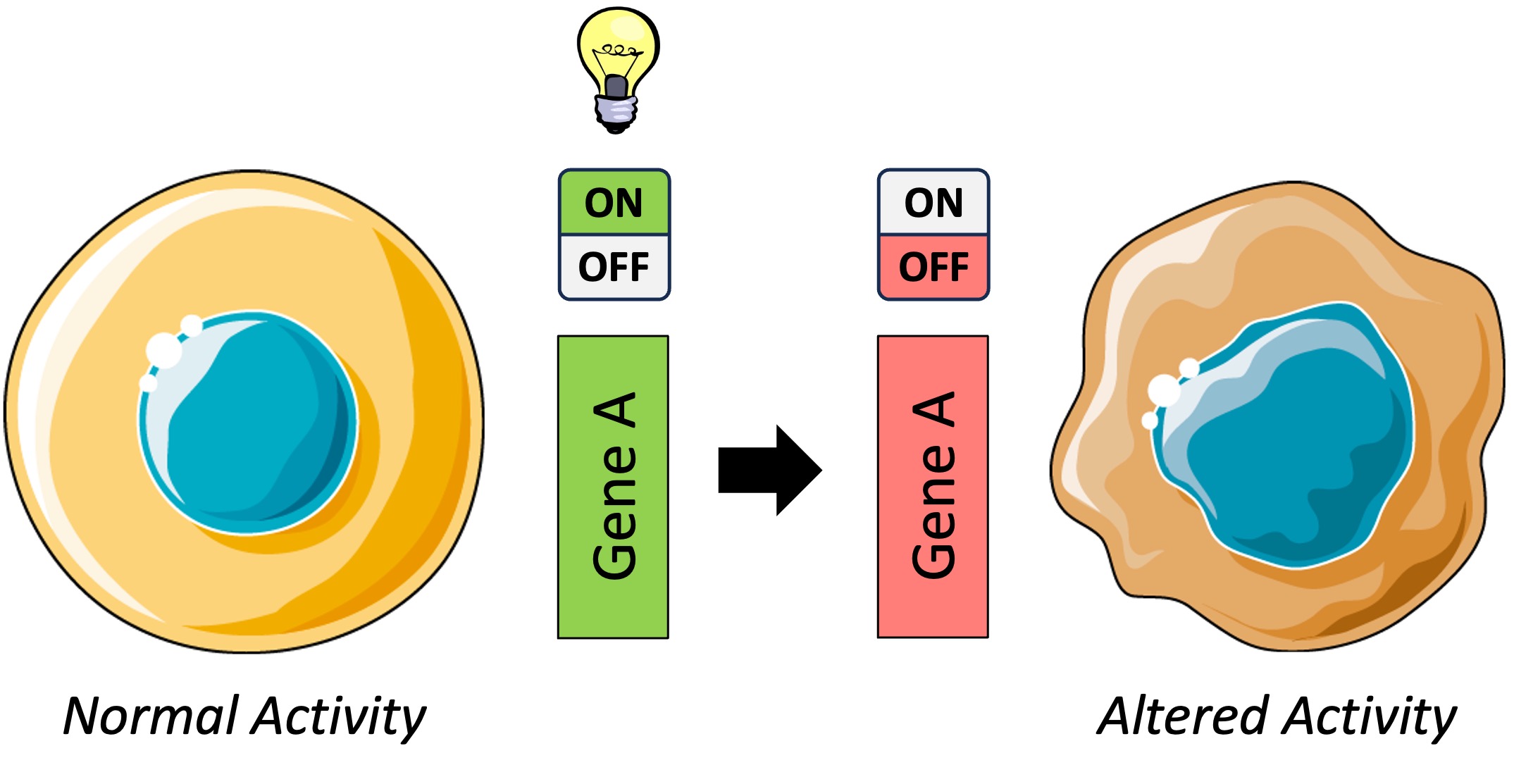
محققان در آزمایشگاه جکسون (JAX)، موسسه Broad در MIT و دانشگاه هاروارد و دانشگاه ییل، از هوش مصنوعی (AI) برای طراحی هزاران سوئیچ DNA جدید استفاده کردهاند که میتوانند به طور دقیق بیان یک ژن را در انواع مختلف سلولها کنترل کنند. روش جدید آنها میتواند بیوتکنولوژی را متحول کند و امکان فعالسازی یا سرکوب دقیق ژنها را در بافتهای خاص فراهم کند.
Ryan Tewhey دانشیار در آزمایشگاه جکسون و یکی از نویسندگان اصلی این تحقیق، میگوید: «ویژگی خاص این عناصر طراحی شده بهطور مصنوعی این است که ویژگیهای مشخصی برای نوع سلول هدفی که برای آن طراحی شدهاند، دارند. این فرصت را برای ما ایجاد میکند که بیان یک ژن را فقط در یک بافت افزایش یا کاهش دهیم بدون اینکه بر بقیه بدن تأثیر بگذارد.»
در سالهای اخیر، فناوریهای ویرایش ژنتیکی و دیگر روشهای درمانی ژن به دانشمندان این امکان را دادهاند که ژنهای داخل سلولهای زنده را تغییر دهند. با این حال، تأثیرگذاری بر ژنها تنها در انواع یا بافتهای خاص سلولی، بهجای سراسر یک ارگانیسم، دشوار بوده است. این مسئله تا حدی به چالشهای موجود در درک سوئیچهای DNA به نام عناصر تنظیمکننده cis (CREs) که بیان و سرکوب ژنها را کنترل میکنند، مربوط میشود.
در مطالعه حاضر، محققان CREهای جدید و بیسابقهای را طراحی کردند و از آنها برای فعالسازی ژنها در سلولهای مغزی، کبدی یا خونی استفاده کردند بدون اینکه این ژنها را در سایر انواع سلولها فعال کنند.
دانشمندان میدانند که هزاران عنصر تنظیمکننده cis (CRE) مختلف در ژنوم انسان وجود دارد که هر کدام نقشهای کمی متفاوت دارند. اما دستور زبان این CREها به سختی قابل درک بوده است، Rodrigo Castro دانشمند محاسباتی در آزمایشگاه Tewhey در جکسون و یکی از نویسندگان اصلی مقاله جدید می گوید: “بدون قوانین واضحی که کنترل کنند هر CRE چه کاری انجام میدهد” او همچنین گفته است: “این محدودیتهایی برای طراحی درمانهای ژنی که تنها بر روی انواع خاصی از سلولها در بدن انسان تأثیر بگذارند، ایجاد میکند.”
استیون ریلی، دانشیار ژنتیک در ییل و یکی از نویسندگان ارشد این مطالعه، میگوید: “این پروژه بهطور اساسی از ما میپرسد: ‘آیا میتوانیم یاد بگیریم که کد این عناصر تنظیمکننده را بخوانیم و بنویسیم؟’ اگر به آن از منظر زبان نگاه کنیم، دستور زبان و نحو این عناصر بهخوبی درک نشده است. بنابراین، ما سعی کردیم روشهای یادگیری ماشین بسازیم که بتوانند یک کد پیچیدهتر از آنچه ما به تنهایی میتوانیم انجام دهیم، یاد بگیرند.”

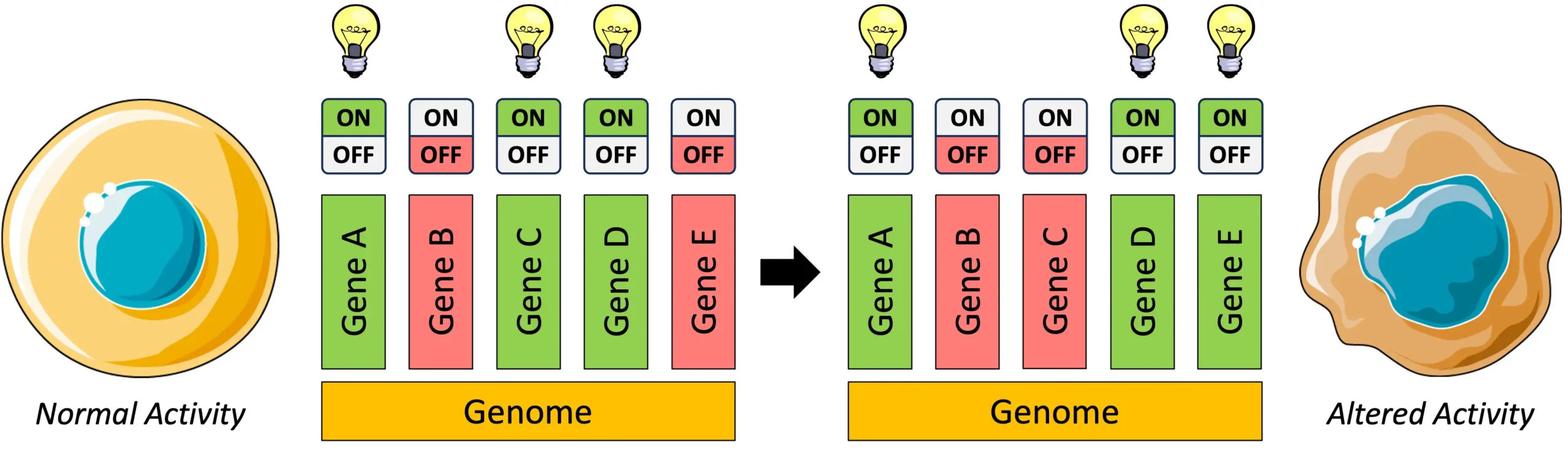
این گروه با استفاده از نوعی هوش مصنوعی به نام یادگیری عمیق، مدلی را با استفاده از صدها هزار توالی DNA از ژنوم انسان که در آزمایشگاه برای فعالیت CRE در سه نوع سلول (خون، کبد و مغز) اندازهگیری شده بود، آموزش دادند. مدل هوش مصنوعی به محققان این امکان را داد تا فعالیت هر توالی را از تقریباً تعداد نامحدودی از ترکیبات ممکن پیشبینی کنند. با تحلیل این پیشبینیها، محققان الگوهای جدیدی در DNA کشف کردند و یاد گرفتند که چگونه دستور زبان توالیهای CRE در DNA بر میزان RNA تولید شده تأثیر میگذارد.
چنین ابزارهایی میتوانند در کنترل دقیق بیان ژن در سلولهای خاص برای اهداف درمانی کاربردهای مهمی داشته باشند.
تیم، شامل پردیس ثابتی، عضو ارشد مطالعه و عضو اصلی در موسسه Broad و استاد در هاروارد، سپس یک پلتفرم به نام CODA (بهینهسازی محاسباتی فعالیت DNA) توسعه دادند که از مدل هوش مصنوعی آنها برای طراحی کارآمد هزاران CRE کاملاً جدید با ویژگیهای درخواست شده، مانند فعالسازی یک ژن خاص در سلولهای کبدی انسان بدون فعالسازی همان ژن در سلولهای خونی یا مغزی انسان استفاده کرد. با ترکیبی تکراری از تحقیقات “تر” و “خشک”، که شامل استفاده از دادههای تجربی برای ساخت و سپس اعتبارسنجی مدلهای محاسباتی بود، محققان توانایی پیشبینی تأثیر بیولوژیکی هر CRE را تقویت و بهبود بخشیدند و طراحی CREهای خاصی که تاکنون در طبیعت دیده نشدهاند، امکانپذیر شد.

Sager Gosai دیگر نویسنده مقاله و پژوهشگر پسادکترا در آزمایشگاه ثابتی، بیان کرد: “CREهای طبیعی، در حالی که فراوان هستند، تنها بخش کوچکی از عناصر ژنتیکی ممکن را نمایندگی میکنند و عملکردشان به دلیل انتخاب طبیعی محدود است.” این ابزارهای هوش مصنوعی پتانسیل عظیمی برای طراحی سوئیچهای ژنتیکی دارند که بهطور دقیق بیان ژن را برای کاربردهای نوین، مانند بیوکارآفرینی و درمان، تنظیم کنند.
Tewhey و همکارانش CREهای مصنوعی طراحی شده با هوش مصنوعی را با اضافه کردن آنها به سلولها و اندازهگیری میزان فعالسازی ژنها در نوع سلول مورد نظر و همچنین توانایی آنها در اجتناب از بیان ژن در سایر سلولها آزمایش کردند. آنها متوجه شدند که CREهای جدید حتی از CREهای طبیعی که با نوع سلولها مرتبط هستند، اختصاصیتر بودند.
Tewhey افزود: “این فناوری راه را برای نوشتن عناصر تنظیمکننده جدید با عملکردهای از پیش تعیین شده هموار میکند.” ابزارهای چنین نوعی برای تحقیقات پایه ارزشمند خواهند بود، اما همچنین میتوانند پیامدهای پزشکی قابل توجهی داشته باشند، جایی که میتوانید از این عناصر برای کنترل بیان ژن در انواع خاص سلولها بهمنظور اهداف درمانی استفاده کنید.
Gosai گفت: «CREهای مصنوعی از نظر معنایی به قدری از عناصر طبیعی متفاوت هستند که پیشبینی اثربخشی آنها دشوار به نظر میرسید. در ابتدا انتظار داشتیم بسیاری از این توالیها در سلولهای زنده نادرست عمل کنند.»
Tewhey و همکارانش دریافتند که CREهای مصنوعی مخصوص سلولهای خاص، شامل توالیهایی برای بیان ژن در نوع خاص سلولها و توالیهایی برای مهار ژن در دیگر سلولها بودند. این گروه چندین CRE مصنوعی را روی ماهی گورخری و موش آزمایش کرد و نتایج مثبتی به دست آورد.
یک CRE توانست پروتئین فلورسنت را در کبد در حال رشد ماهی فعال کند، بدون اینکه در مناطق دیگر فعال شود!
Tewhey بیان کرد: «این فناوری راه را برای ایجاد عناصر تنظیمی جدید با عملکردهای از پیش تعریفشده هموار میکند. چنین ابزارهایی میتوانند در تحقیقات پایه و همچنین در کنترل دقیق بیان ژن در سلولهای خاص برای اهداف درمانی کاربردهای مهمی داشته باشند.»
منبع: وبسایت genengnews